Перейти к:
Возможности и достижения использования массового параллельного секвенирования в диагностике наследственных заболеваний с поражением нервной системы
https://doi.org/10.17749/2077-8333/epi.par.con.2023.127
Аннотация
Вклад генетических факторов в развитие неврологических заболеваний известен давно, и большинство достижений при использовании молекулярно-генетических технологий заключалось в выявлении генов, связанных с патологией развития нервной системы. Цель настоящего обзора – показать возможности и достижения использования метода массового параллельного секвенирования в диагностике заболевания с поражением нервной системы. В настоящее время технология секвенирования нового поколения (англ. next generation sequencing, NGS) в генетической диагностике эпилепсии включает использование целевых генных панелей, секвенирование всего экзома или генома. Интеграция генетического и геномного анализа в клиническую практику для установления четких молекулярных диагнозов для ранее не диагностированных пациентов позволит избежать дальнейшей ненужной диагностики, а следовательно, приведет к большей экономии расходов на здравоохранение с течением времени. Применение биомаркеров будет способствовать прогнозированию исхода болезни и принятию решения о терапии.
Ключевые слова
Для цитирования:
Кожанова Т.В. Возможности и достижения использования массового параллельного секвенирования в диагностике наследственных заболеваний с поражением нервной системы. Эпилепсия и пароксизмальные состояния. 2023;15(1):44-52. https://doi.org/10.17749/2077-8333/epi.par.con.2023.127
For citation:
Kozhanova T.V. Opportunities and achievements of using massive parallel sequencing in the diagnosis of neurodevelopmental diseases. Epilepsy and paroxysmal conditions. 2023;15(1):44-52. (In Russ.) https://doi.org/10.17749/2077-8333/epi.par.con.2023.127
ВВЕДЕНИЕ / INTRODUCTION
Вклад генетических факторов в развитие неврологических заболеваний известен давно, и большинство достижений при использовании молекулярно-генетических технологий заключалось в выявлении генов, связанных с патологией развития нервной системы [1]. В геноме человека около 20 тыс. различных генов, более 80% из них активно экспрессируются в головном мозге [2]. Согласно базе данных наследственных заболеваний Online Mendelian Inheritance in Man (OMIM) для 7 тыс. генетических нарушений установлена молекулярная основа, при этом 40% заболеваний связаны с патологией нервной системы. Однако до внедрения массового параллельного секвенирования (секвенирования нового поколения – англ. next-generation sequencing, NGS) фенотипическая и генотипическая гетерогенность и использование низкопроизводительных технологий для генетического тестирования (прежде всего, секвенирование по Сэнгеру) указывали на то, что большинство пациентов не получали молекулярного диагноза, расчета риска повторения заболевания для членов семьи и возможности таргетной терапии на основе понимания механизмов болезни [3].
С момента появления метода секвенирования экзома в качестве клинического диагностического теста он стал рутинно использоваться у пациентов с подозрением на редкие генетические нарушения [2]. Во многих лабораториях также начали внедрять генные панели, или тесты таргетного секвенирования, на основе NGS, заменяя секвенирование по Сэнгеру, количественную полимеразную цепную реакцию и мультиплексную лиганд-зависящую амплификацию, поскольку NGS является более экономичным, оптимизированным методом и позволяет легко увеличить количество анализируемых таргетных генов. Технология не только позволила гораздо шире и быстрее устанавливать клинический диагноз [3–5], но также помогла исследователям идентифицировать новые, ранее неизвестные неврологические расстройства [6]. Таким образом, использование технологии, способной «сканировать» весь генетический ландшафт пациента с целью выявления критически значимой клинической информации, способствовало развитию персонализированной медицины и прецизионной диагностики и терапии.
Цель – показать возможности и достижения использования технологии массового параллельного секвенирования в диагностике заболевания с поражением нервной системы.
МАТЕРИАЛ И МЕТОДЫ / MATERIAL AND METHODS
Поиск статей осуществлялся в англоязычных базах данных (PubMed/MEDLINE) с использованием ключевых слов и словосочетаний на английском языке: “genetics of epilepsy”, “epileptic encephalopathy”, “genetics of autism”, “next-generation sequencing”, “genetic diagnoses in epilepsy”, “epilepsy associated genes”, “whole exome sequencing”. В период с 1 марта 2022 г. по 28 апреля 2022 г. были проанализированы полнотекстовые публикации оригинальных исследований на английском языке. В базах данных найдено 563 статьи. После удаления дублирующих текстов, тщательно проведенного скрининга, оценки полнотекстовых статей на приемлемость в соответствии с критериями отбора в анализ включено 47 научных публикаций. Приоритет был отдан исследованиям за период 2017–2022 гг.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
История генетического тестирования в диагностике эпилепсии / History of genetic testing in the diagnosis of epilepsy
За последние 20 лет совершен значительный прорыв в понимании молекулярных механизмов болезней человека. Эпилепсия не стала исключением. Являясь одним из распространенных неврологических заболеваний, она характеризуется рецидивирующими неспровоцированными судорогами, которые поражают людей любого возраста, пола, расы и географического местоположения. Почти у 50 млн чел. во всем мире диагностирована эпилепсия (по данным Всемирной организации здравоохранения, около 1% населения), при этом показатель распространенности варьирует от 5 до 10 случаев на 1 тыс. чел.
Эпилепсия – неврологическое заболевание, которое не только снижает качество жизни пациента, но и становится тяжелым грузом для родственников больного [7][8]. Одной из тяжелых форм проявления эпилепсии является энцефалопатия развития, или эпилептическая энцефалопатия. Она относится к группе моногенных форм эпилепсии, манифестирует на первом году жизни и характеризуется частыми полиморфными судорогами, резистентными к противосудорожной терапии, выраженной задержкой психомоторного и речевого развития, а также высоким уровнем инвалидизации и смертности [8]. Эпилепсия – основное заболевание в педиатрической неврологии в связи с тем, что до 80% случаев судорог дебютируют в детском и подростковом возрасте. До периода активного развития и внедрения технологий генетического тестирования уже предполагалось, что в основе развития данного заболевания, возможно, лежит повреждение генома. Были описаны семейные случаи развития болезни [9–13]. В настоящее время уже известно, что с развитием эпилепсии ассоциировано более 1 тыс. генов (так называемые моногенные формы, когда вариант нуклеотидной последовательности в одном гене приводит к заболеванию, и полигенные формы – вовлеченность в патологический процесс нескольких генов) [8][14].
Следует отметить, что десятилетие назад врачи сталкивались с большими трудностями в отношении генетической диагностики эпилепсии. Во-первых, это связано с тем, что бо́льшая часть генов еще не была идентифицирована, во-вторых, те гены, структура которых была установлена, имеют больший размер, что обусловливает значительные экономические затраты на секвенирование их по отдельности.
Спустя годы удалось добиться значительного повышения эффективности и снижения экономических затрат благодаря развитию и внедрению в клиническую практику массового параллельного секвенирования, или технологии NGS. Кроме того, применение данного метода способствовало открытию новых генов, ассоциированных не только с моногенными формами эпилепсии, но и с другими наследственными заболеваниями, где ведущим симптомом поражения нервной системы являются судороги, благодаря возможности одновременного тестирования нуклеотидной последовательности большого количества генов [15][16]. В работе, выполненной J. Wang et al. (2017 г.), было выявлено, что с развитием судорог ассоциированы более 900 генов: моногенные формы эпилепсии (первичное заболевание), редкие генетические синдромы и наследственные болезни обмена с преимущественным поражением нервной системы, где одним из ведущих симптомов являются судороги [17].
Другая причина, которую также необходимо принимать во внимание при проведении медико-генетического консультирования семьи с больным эпилепсией, – наличие мозаичных вариантов нуклеотидной последовательности в известных генах (например, SCN1A и SLC6A1) [18][19]. Так, при обследовании пробандов на 9 генов (CDKL5, GABRA1, GABRG2, GRIN2B, KCNQ2, MECP2, PCDH19, SCN1A и SCN2A), варианты нуклеотидной последовательности в которых наиболее часто приводят к эпилепсии, M. Stosser et al. (2017 г.) показали, что частота мозаицизма достигала 3,5% [20]. Однако, несмотря на развитие технологии генетического тестирования, проблема обнаружения мозаицизма остается актуальной [20], т.к. он не ограничен только изменениями в одном гене или в митохондриальной ДНК, но также может наблюдаться при хромосомных нарушениях [21].
В дальнейшем в ряде работ было обосновано, что генетическое тестирование должно быть одним из основных исследований в диагностике эпилепсии [16]. Таким образом, массовое параллельное секвенирование не только является современной технологией генетического тестирования эпилепсии, но и имеет высокий потенциал для выявления семейных вариантов нуклеотидной последовательности и вариантов, возникших впервые (de novo).
Виды массового параллельного секвенирования / Types of massive parallel sequencing
В настоящее время в генетической диагностике заболеваний с преимущественным поражением нервной системы технология массового параллельного секвенирования применяется в виде использования отдельных панелей генов (таргетные панели), секвенирования клинического экзома, полноэкзомного секвенирования (ПЭС) и полногеномного секвенирования (ПГС).
Таргетные панели
Таргетные панели позволяют секвенировать определенную группу клинически значимых генов и проводить фенотип-генотипические корреляции [22]. Однако из-за увеличивающегося количества вновь открытых генов вместо них все чаще применяются ПЭС и ПГС. Генные панели имеют свои преимущества и недостатки. При использовании панели генов существует возможность значительного увеличения покрытия при сохранении низкой стоимости секвенирования. Но нельзя идентифицировать новые гены, могут выявляться варианты, ошибочно принимаемые за причину заболевания. Чем больше панель, тем меньше разница в стоимости с ПЭС. Кроме того, при применении панели генов отсутствуют «неожиданные» находки.
Клинический экзом
Клинический экзом – исследование экзонов генов, ассоциированных с заболеваниями. Стоимость его ниже, чем при использовании ПЭС и ПГС. В рамках применения клинического экзома есть возможность анализа как всех данных, так и данных в рамках панелей генов. Однако при этом невозможно досеквенировать все непокрытые экзоны. В анализ данных не включены интроны и регуляторные области. Покрытие менее равномерное, чем при ПГС.
Полноэкзомное секвенирование
ПЭС получило широкое применение в клинической практике для генетической диагностики. Технология направлена на секвенирование белок-кодирующей области генома, которая составляет приблизительно 1–2%. Благодаря использованию ПЭС выявляемость вариантов, связанных с заболеванием, может достигать 85% [23–25].
Данный метод особенно эффективен в выявлении вариантов в генах при фенотипически вариабельных состояниях и используется для молекулярной диагностики таких неврологических нарушений, как эпилепсия, энцефалопатия развития и эпилептическая энцефалопатия, патология интеллектуального дефицита, и других нарушений развития нервной системы, например мозжечковых атаксий (рис. 1) [3–5][26]. Когда фенотипы болезни, которые бесспорно являются генетическими, должным образом идентифицированы, валидированы, а затем проанализированы с использованием секвенирования экзома, примерно 40% пациентов можно поставить молекулярный диагноз [27], а для некоторых заболеваний данный показатель может достигать 94% [27][28].
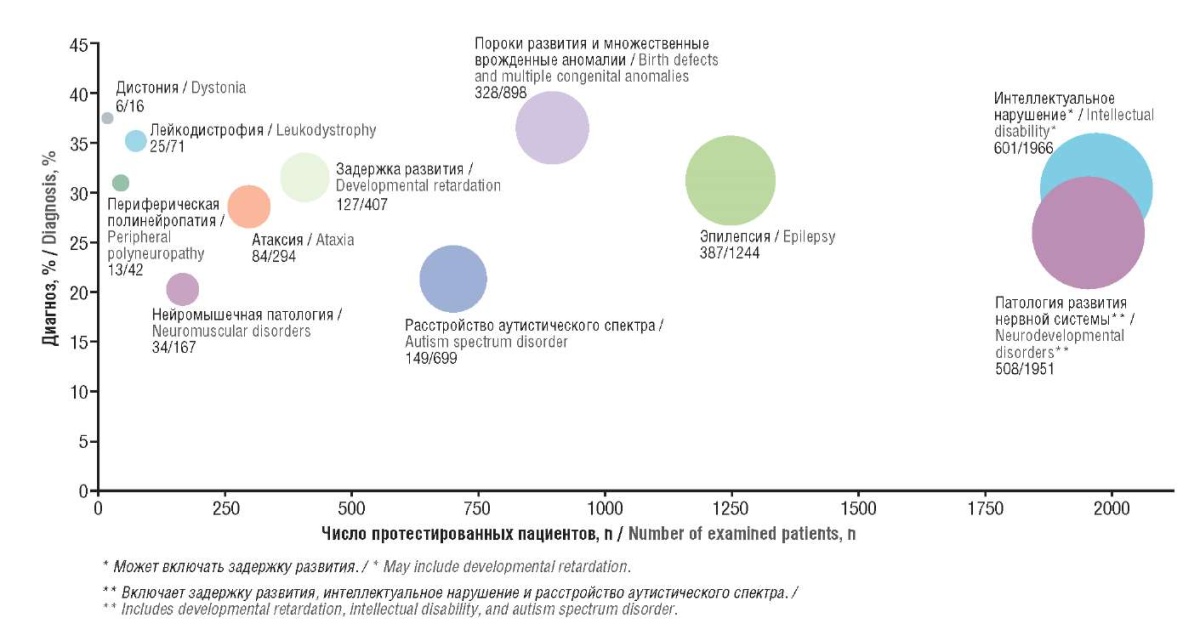
Рисунок 1. Доли пациентов, у которых на молекулярном уровне диагностированы различные неврологические заболевания с помощью полноэкзомного секвенирования (общее количество пациентов указано для каждого неврологического расстройства и отражено в относительном размере для каждой точки данных) [26]
Figure 1. Proportion of patients diagnosed at the molecular level with various neurological diseases by using whole exome sequencing (total number of patients indicated for each neurological disorder and reflected in relative size for each data point) [26]
Секвенирование экзома в настоящее время считается методом выбора при диагностике моногенных неврологический нарушений – как правило, в сочетании с другими технологиями для выявления вариаций числа копий (микроделеций и микродупликаций), например хромосомный микроматричный анализ. Хотя ПЭС и хромосомные микроматрицы в настоящее время уже могут быть заменены ПГС.
Применение технологии массового параллельного секвенирования в последнее десятилетие не только расширило наше знание об этиологии и патогенезе эпилепсии, энцефалопатии развития и эпилептической энцефалопатии (согласно базе данных OMIM выделяют 109 типов), дало возможность проследить генотип-фенотипические корреляции между типом, локализацией вариантов и тяжестью клинических проявлений, но и привело к значительному успеху в выборе персонифицированного лечения больных (таргетная противосудорожная терапия). Так, например, у пациентов с подтвержденной пиридоксин-зависимой эпилепсией (мутация в гене ALDH7A1) положительный эффект обеспечивается при назначении терапии препаратами витамина B6, при синдроме дефицита транспортера глюкозы 1-го типа (мутация в гене SLC2A1) исправление определенных метаболических дефектов и снижение (или полное устранение) судорог достигается использованием специализированной кетогенной диеты [24][25].
Расстройство аутистического спектра (РАС) часто связано с эпилептической энцефалопатией у детей. Однако ранняя диагностика РАС у детей с эпилепсией затруднена из-за вариабельности клинических фенотипов. Было высказано предположение об общих как биологических, так и молекулярных механизмах РАС и эпилепсии, например при комплексе туберозного склероза. Гипотетические объяснения, подтверждающие взаимосвязь двух состояний, включают аномалии роста синапсов, дисбаланс возбуждения/торможения нейронов и аномальную синаптическую пластичность [29]. В текущее десятилетие возрос интерес к пониманию генетических причин, приводящих к РАС, нарушениям интеллектуального развития и эпилепсии. В значительной степени это обусловлено внедрением современных геномных технологий, которые помогают идентифицировать причину заболевания и на основании полученных данных проводить персонифицированное лечение таких пациентов. При исследовании генетической основы было выделено множество генов, связанных с аномалиями ионных каналов, синаптической функции и структуры, а также регуляторами транскрипции пути mTOR [29].
Очень интересна ассоциация между эпилепсией детства и сопутствующим РАС. Эпилепсия может привести к РАС или, наоборот, патология головного мозга, лежащая в основе РАС, может предрасполагать к судорогам. Общий биологический механизм эпилепсии детства и РАС включает подгруппу генов эпилепсии и аутизма, которые имеют схожие фенотипические проявления и биологические функции. Инфантильные судороги при моногенных эпилептических энцефалопатиях наиболее часто ассоциируются с РАС. ПГС- и ПЭС-исследования пролили свет на генетические причины как эпилепсии детства, так и РАС [28].
На рисунке 2 представлены гены, которые наиболее часто ассоциируются с развитием эпилепсии и РАС, сгруппированные согласно различным механизмам действия [29–31].
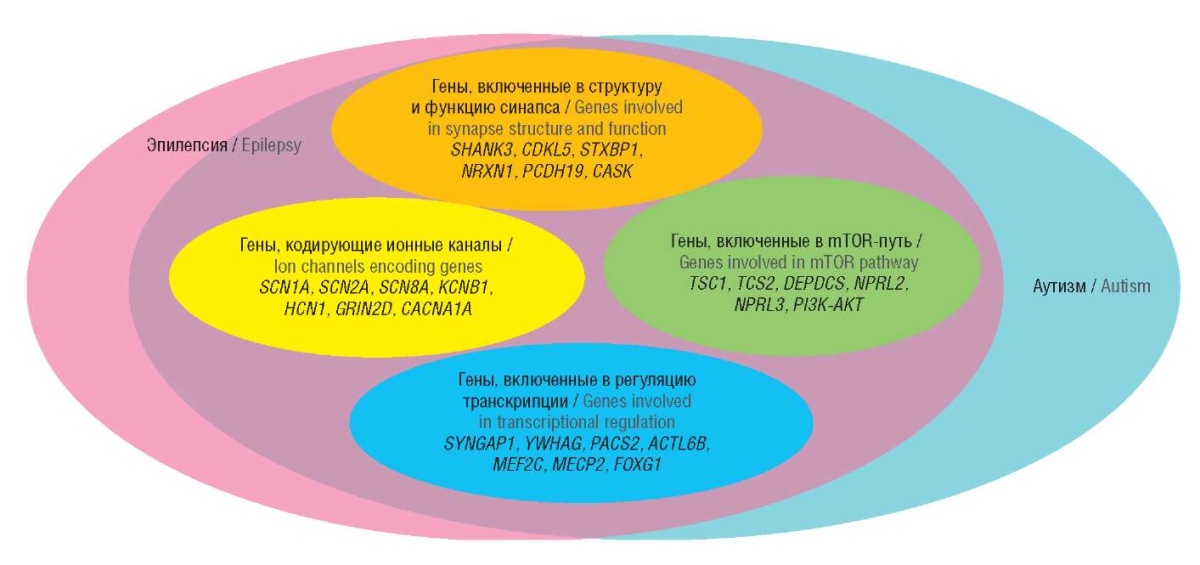
Рисунок 2. Мутации в генах, лежащих в основе эпилепсии и аутизма [29–31]
Figure 2. Mutations in epilepsy and autism underlying genes [29–31]
У пациента с неспецифическим фенотипом без предварительного диагноза диагностический показатель ПЭС может достигать 25–30% [32–34].
Частота развития эпилепсии среди пациентов с РАС составляет до 23% и возрастает с тяжестью заболевания [35]. Примерно у 40% больных с ранее необъяснимой задержкой развития или интеллектуальным дефицитом до внедрения ПЭС этиологический диагноз мог быть установлен только с помощью клинического обследования и последующих специфических диагностических тестов (ДНК-диагностика), хромосомного микроматричного анализа (ХМА) и скрининга на наследственные нарушения обмена [36–38]. В последующих исследованиях было показано, что эффективность выявления генетической причины РАС, интеллектуального дефицита в сочетании с эпилепсией с помощью ПЭС достигает 70% [39], в отличие от других методов (исследование кариотипа – 5–10%, ХМА – 15–20%) [31][39].
Помимо этого есть много причин, по которым ПЭС может не идентифицировать генетическую причину. Некоторые из них носят технический характер – как правило, из-за неполной или неоднородной глубины покрытия, иногда при наличии геномных участков, которые трудно секвенировать (например, области, богатые гуанином и цитозином, высоко повторяющиеся элементы и псевдогены). Кроме того, некоторые типы вариантов, такие как экспансии тринуклеотидных повторов, сложные вариации числа копий или другие варианты, вызывающие структурные аномалии в геноме, трудно идентифицировать, и поэтому они требуют специализированного тестирования или дополнительных биоинформационных паплайнов (алгоритмов) [40]. Варианты в некодирующих последовательностях генома и соматический или герминальный мозаицизм обычно не выявляются с помощью ПЭС. В конечном счете недостаточная клиническая информация и невозможность идентифицировать все известные гены, связанные с заболеванием, могут быть наиболее частыми причинами, препятствующими диагностике. Специфические расстройства, для которых эффективность ПЭС все еще не доказана, включают болезнь Паркинсона, болезнь Альцгеймера и цереброваскулярные расстройства [40].
Причины отрицательных результатов ПЭС:
- варианты находятся за пределами разрешающей способности метода (интронные варианты, экспансия тринуклеотидных повторов, делеции/дупликации;
- соматические варианты;
- многофакторная этиология заболевания;
- варианты находятся в гене, функция которого неизвестна и ассоциация с заболеванием не установлена.
Полногеномное секвенирование
Другой методикой массового параллельного секвенирования, уже активно внедряющейся в клиническую практику, является ПГС, которое позволяет идентифицировать любую поломку в геноме – от точечных вариантов в кодирующей части гена (экзоне), некодирующей (интроне) и регуляторной области до хромосомных микроперестроек [22][34]. Нетранслируемые регионы, которые могут играть регуляторную роль (например, некодирующие РНК или сайты связывания транскрипции), вместе с потенциальными сайтами кодирования белка охватывают 99% генома [41][42]. В настоящее время не совсем ясно влияние таких вариантов на геном, процессы транскрипции и трансляции. Авторами научных публикаций по данной проблеме было показано, что они могут оказывать влияние на уровни экспрессии или сплайсинга матричной РНК. Растущее число этих вариантов имеет прогностическую и терапевтическую значимость с точки зрения персонализированной медицины.
Некоторые лаборатории начали предлагать ПГС вместо или в дополнение к ПЭС. Полный геном имеет примерно 4,5–5,0 млн однонуклеотидных вариантов и вариантов «вставка/делеция» на образец [33][43]. После фильтрации из распространенных и, вероятно, доброкачественных вариантов около 400 тыс. обычно остаются для интерпретации в контексте клинической информации. Значение ПГС в неврологической практике оценивалось в рамках проекта по 100 тыс. геномам – крупнейшего исследования, направленного на использование геномики для улучшения помощи больным.
К преимуществам ПГС по сравнению с ПЭС относится возможность идентифицировать вариации числа копий с более высокой чувствительностью и лучшим разрешением [44]. Другими достоинствами метода являются более равномерная глубина покрытия и высокий потенциал для выявления других типов вариантов, таких как экспансии тринуклеотидных повторов, которые часто служат причинами неврологических заболеваний, например болезни Гентингтона или бокового амиотрофического склероза [45].
Проблемы при проведении секвенирования / Problems with sequencing
Несмотря на столь высокие достижения в области молекулярной диагностики и получения большого количества геномной информации, врач-генетик при трактовке результатов секвенирования экзома и генома сталкивается с определенными проблемами:
- обнаружение ранее не описанных вариантов нуклеотидной последовательности, функциональная значимость которых еще не известна;
- выявление вариантов в двух генах, ответственных за возникновение заболеваний одной группы, сходных по клиническим проявлениям;
- обнаружение варианта в гетерозиготном состоянии в гене, ответственном за аутосомно-рецессивное заболевание, клинические проявления которого сходны с таковыми у больного (второй вариант в интроне? крупная делеция? экспансия нуклеотидных повторов?);
- установление цис-/транс-положения двух вариантов в гене, ответственном за возникновение аутосомнорецессивного заболевания;
- неполная пенетрантность и варьирующая экспрессивность гена, ответственного за возникновение аутосомно-доминантного заболевания, приводящие к отсутствию или наличию минимальных клинических проявлений у носителей патогенного варианта, что требует их тщательного обследования;
- гонадный мозаицизм, уровень которого не всегда известен.
Кроме того, получение отрицательного результата при проведении ПЭС и ПГС не исключает полностью генетической патологии, особенно при фенотипе, указывающем на возможную поломку в геноме. Наука не стоит на месте, ежегодно появляется большое количество информации о новых генах и их взаимосвязях с заболеваниями. Поэтому спустя 2–3 года возможно провести повторный анализ полученных данных секвенирования.
Таргетная терапия моногенных форм эпилепсии / Targeted therapy of monogenic forms of epilepsy
Массовое параллельное секвенирование позволяет не только быстро и с высокой эффективностью поставить окончательный молекулярный диагноз, но и выявить вариант нуклеотидной последовательности в конкретном гене, что даст возможность врачу назначить разработанный орфанный препарат, персонифицировать профилактику и применение препаратов, в частности при эпилепсии. Таблица 1 составлена автором на основании последней публикации по таргетной терапии моногенных форм эпилепсии [46].
Таблица 1. Возможности таргетной терапии при эпилепсии (адаптировано из [46])
Table 1. Рossibilities of targeted therapy in epilepsy (adapted from [46])

ЗАКЛЮЧЕНИЕ / CONCLUSION
Таким образом, в генетике и диагностике неврологических заболеваний началась новая эра тесного сотрудничества исследователей и клиницистов. В настоящее время наблюдается интенсивный рост в области доказательной терапии, особенно для генетически и фенотипически гетерогенной группы такой тяжелой формы эпилепсии, как эпилептические энцефалопатии.
С развитием технологий массового параллельного секвенирования основными инструментами в руках клинических специалистов для выявления причины заболевания стали ПЭС и ПГС. Определение особенностей клинических проявлений у больных с отдельными генетическими и фенотипическими вариантами энцефалопатии развития и эпилептической энцефалопатией, в свою очередь, позволит оптимизировать процесс их молекулярно-генетической диагностики и повысить эффективность медико-генетического консультирования семей с отягощенным семейным анамнезом. Как только вариант в гене моногенного или полигенного заболевания как причина патологии четко определяется, соответствующая таргетная терапия может быть направлена на устранение молекулярного дефекта в данном гене или компенсацию нарушения молекулярного пути.
В ближайшем будущем увеличивающийся объем информации по выявлению мутаций не только в кодирующей части генома, но и в интронных и регуляторных регионах позволит в клинической практике перейти от рутинного использования ПЭС к применению ПГС, когда в одном тесте возможно определять точечные варианты, вариации числа копий, экспансии тринуклеотидных повторов, а также секвенировать митохондриальную ДНК, исключая тем самым большинство известных наследственных заболеваний с преимущественным поражением нервной системы. Полученные данные помогут не только найти тот самый каузативный вариант нуклеотидной последовательности, поставив финальную точку в длительной одиссее для пациента, но и углубить наше знание патогенеза заболевания.
Однако нельзя забывать, что до назначения и начала и проведения любого генетического исследования пациенты и члены их семей обязательно должны быть проинформированы направляющим врачом об этических проблемах, которые могут возникать из результатов генетического тестирования. Особенно это касается так называемых вторичных находок, когда в процессе интерпретации данных выявляется мутация в гене, не связанном с основным направляющим диагнозом, но пациенту необходимо сообщить об этом в целях профилактики осложнения заболевания и возможности назначения ранней терапии. Также важно поставить в известность больных и их родственников о других аспектах, таких как технические ограничения метода, правовые нормы и расходы на генетическое тестирование.
Список литературы
1. Pihlstrom L., Wiethoff S., Houlden H. Genetics of neurodegenerative diseases: an overview. Handb Clin Neurol. 2017; 145: 309–23. https://doi.org/10.1016/B978-0-12-802395-2.00022-5.
2. Fogel B.L. Genetic and genomic testing for neurologic disease in clinical practice. Handb Clin Neurol. 2018; 147: 11–22. https://doi.org/10.1016/B978-0-444-63233-3.00002-6.
3. Fogel B.L., Lee H., Deignan J.L., et al. Exome sequencing in the clinical diagnosis of sporadic or familial cerebellar ataxia. JAMA Neurol. 2014; 71 (10): 1237–46. https://doi.org/10.1001/jamaneurol.2014.1944.
4. Pyle A., Smertenko T., Bargiela D., et al. Exome sequencing in undiagnosed inherited and sporadic ataxias. Brain. 2015; 138 (Pt. 2): 276–83. https://doi.org/10.1093/brain/awu348.
5. Farwell K.D., Shahmirzadi L., El-Khechen D., et al. Enhanced utility of family-centered diagnostic exome sequencing with inheritance modelbased analysis: results from 500 unselected families with undiagnosed genetic conditions. Genet Med. 2015; 17 (7): 578–86. https://doi.org/10.1038/gim.2014.154.
6. Boycott K.M., Rath A., Chong J.X., et al. International cooperation to enable the diagnosis of all rare genetic diseases. Am J Hum Genet. 2017; 100 (5): 695–705. https://doi.org/10.1016/j.ajhg.2017.04.003.
7. Fisher R.S., van Emde Boas W., Blume W., et al. Epileptic seizures and epilepsy: definitions proposed by the International League Against Epilepsy (ILAE) and the International Bureau for Epilepsy (IBE). Epilepsia. 2005; 46 (4): 470–2. https://doi.org/10.1111/j.0013-9580.2005.66104.x.
8. Moller R.S., Dahl H.A., Helbig I. The contribution of next generation sequencing to epilepsy genetics. Expert Rev Mol Diagn. 2015; 15 (12): 1531–8. https://doi.org/10.1586/14737159.2015.1113132.
9. Annegers J.F., Hauser W.A., Anderson V.E., Kurland L.T. The risks of seizure disorders among relatives of patients with childhood onset epilepsy. Neurology. 1982; 32 (2): 174–9. https://doi.org/10.1212/WNL.32.2.174.
10. Scheffer I.E., Berkovic S.F. Generalized epilepsy with febrile seizures plus. A genetic disorder with heterogeneous clinical phenotypes. Brain. 1997; 120 (Pt. 3): 479–90. https://doi.org/10.1093/brain/120.3.479.
11. Jallon P., Loiseau P., Loiseau J. Newly diagnosed unprovoked epileptic seizures: presentation at diagnosis in CAROLE Study. Epilepsia. 2001; 42 (4): 464–75. https://doi.org/10.1046/j.1528-1157.2001.31400.x.
12. Myers C.T., Mefford H.C. Advancing epilepsy genetics in the genomic era. Genome Med. 2015; 7 (1): 91. https://doi.org/10.1186/s13073-015-0214-7.
13. Myers K.A., Johnstone D.L., Dyment D.A. Epilepsy genetics: current knowledge, applications, and future directions. Clin Genet. 2019; 95 (1): 95-111. https://doi.org/10.1111/cge.13414.
14. Poduri A., Lowenstein D. Epilepsy genetics – past, present, and future. Curr Opin Genet Dev. 2011; 21 (3): 325–32. https://doi.org/10.1016/j.gde.2011.01.005.
15. Dunn P., Albury C.L., Maksemous N., et al. Next generation sequencing methods for diagnosis of epilepsy syndromes. Front Genet. 2018; 9: 20. https://doi.org/10.3389/fgene.2018.00020.
16. Berkovic S.F. Genetics of epilepsy in clinical practice. Epil Curr. 2015; 15 (4): 192–6. https://doi.org/10.5698/1535-7511-15.4.192.
17. Wang J., Lin Z.J., Liu L., et al. Epilepsy-associated genes. Seizure. 2017; 44: 11–20. https://doi.org/10.1016/j.seizure.2016.11.030.
18. Shi Y.W., Yu M.J., Long Y.S., et al. Mosaic SCN1A mutations in familial partial epilepsy with antecedent febrile seizures. Genes Brain Behav. 2012; 11 (2): 170–6. https://doi.org/10.1111/j.1601-183X.2011.00756.x.
19. Halvorsen M., Petrovski S., Shellhaas R., et al. Mosaic mutations in earlyonset genetic diseases. Genet Med. 2016; 18 (7): 746–9. https://doi.org/10.1038/gim.2015.155.
20. Stosser M.B., Lindy A.S., Butler E., et al. High frequency of mosaic pathogenic variants in genes causing epilepsy-related neurodevelopmental disorders. Genet Med. 2018; 20 (4): 403–10. https://doi.org/10.1038/gim.2017.114.
21. Gajecka M. Unrevealed mosaicism in the next-generation sequencing era. Mol Genet Genomics. 2016; 291 (2): 513–30. https://doi.org/10.1007/s00438-015-1130-7.
22. Poduri A., Sheidley B.R., Shostak S., Ottman R. Genetic testing in the epilepsies-developments and dilemmas. Nat Rev Neurol. 2014; 10 (5): 293–9. https://doi.org/10.1038/nrneurol.2014.60.
23. Choi M., Scholl U.I., Ji W., et al. Genetic diagnosis by whole exome capture and massively parallel DNA sequencing. Proc Natl Acad Sci. USA. 2009; 106 (45): 19096–101. https://doi.org/10.1073/pnas.0910672106.
24. Hunt A.D. Jr., Stokes J. Jr., Mc C.W., Stroud H.H. Pyridoxine dependency: report of a case of intractable convulsions in an infant controlled by pyridoxine. Pediatrics. 1954; 13 (2): 140–5.
25. Mills P.B., Struys E., Jakobs C., et al. Mutations in antiquitin in individuals with pyridoxine-dependent seizures. Nat Med. 2006; 12 (3): 307–9. https://doi.org/10.1038/nm1366.
26. Rexach J., Lee H., Martinez-Agosto J.A., et al. Clinical application of nextgeneration sequencing to the practice of neurology. Lancet Neurol. 2019; 18 (5): 492–503. https://doi.org/10.1016/S1474-4422(19)30033-X.
27. Akizu N., Silhavy J.L., Rosti R.O., et al. Mutations in CSPP1 lead to classical Joubert syndrome. Am J Hum Genet. 2014; 94 (1): 80–6. https://doi.org/10.1016/j.ajhg.2013.11.015.
28. Vilboux T., Doherty D.A., Glass I.A., et al. Molecular genetic findings and clinical correlations in 100 patients with Joubert syndrome and related disorders prospectively evaluated at a single center. Genet Med. 2017; 19 (8): 875–82. https://doi.org/10.1038/gim.2016.204.
29. Specchio N., Di Micco V., Trivisano M., et al. The epilepsy-autism spectrum disorder phenotype in the era of molecular genetics and precision therapy. Epilepsia. 2022; 63 (1): 6–21. https://doi.org/10.1111/epi.17115.
30. Yang Y., Muzny D.M., Reid J.G., et al. Clinical whole-exome sequencing for the diagnosis of mendelian disorders. N Engl J Med. 2013; 369 (16): 1502–11. https://doi.org/10.1056/NEJMoa1306555.
31. Yang Y., Muzny D.M., Xia F., et al. Molecular findings among patients referred for clinical whole-exome sequencing. JAMA. 2014; 312 (18): 1870–9. https://doi.org/10.1001/jama.2014.14601.
32. Jiang Y.H., Yuen R.K., Jin X., et al. Detection of clinically relevant genetic variants in autism spectrum disorder by whole-genome sequencing. Am J Hum Genet. 2013; 93 (2): 249–63. https://doi.org/10.1016/j.ajhg.2013.06.012.
33. Gilissen C., Hehir-Kwa J.Y., Thung D.T., et al. Genome sequencing identifies major causes of severe intellectual disability. Nature. 2014; 511 (7509): 344–7. https://doi.org/10.1038/nature13394.
34. Stavropoulos D.J., Merico D., Jobling R., et al. Whole-genome sequencing expands diagnostic utility and improves clinical management in paediatric medicine. NPJ Genom Med. 2016; 1: 15012. https://doi.org/10.1038/npjgenmed.2015.12.
35. Robertson J., Hatton C., Emerson E., Baines S. Prevalence of epilepsy among people with intellectual disabilities: a systematic review. Seizure. 2015; 29: 46–62. https://doi.org/10.1016/j.seizure.2015.03.016.
36. Leonard H., Wen X. The epidemiology of mental retardation: challenges and opportunities in the new millennium. Ment Retard Dev Disabil Res Rev. 2002; 8 (3): 117–34. https://doi.org/10.1002/mrdd.10031.
37. Ropers H.H. Genetics of early onset cognitive impairment. Annu Rev Genomics Hum Genet. 2010; 11: 161–87. https://doi.org/10.1146/annurevgenom-082509-141640.
38. Topper S., Ober C., Das S. Exome sequencing and the genetics of intellectual disability. Clin Genet. 2011; 80 (2): 117–26. https://doi.org/10.1111/j.1399-0004.2011.01720.x.
39. Orsini A., Zara F., Striano P. Recent advances in epilepsy genetics. Neurosci Lett. 2018; 667: 4–9. https://doi.org/10.1016/j.neulet.2017.05.014.
40. Mandelker D., Schmidt R.J., Ankala A., et al. Navigating highly homologous genes in a molecular diagnostic setting: a resource for clinical next-generation sequencing. Genet Med. 2016; 18 (12): 1282–9. https://doi.org/10.1038/gim.2016.58.
41. Chrystoja C.C., Diamandis E.P. Whole genome sequencing as a diagnostic test: challenges and opportunities. Clin Chem. 2014; 60 (5): 724–33. https://doi.org/10.1373/clinchem.2013.209213.
42. Lohmann K., Klein C. Next generation sequencing and the future of genetic diagnosis. Neurotherapeutics. 2014; 11 (4): 699–707. https://doi.org/10.1007/s13311-014-0288-8.
43. Auton A., Brooks L.D., Durbin R.M., et al. A global reference for human genetic variation. Nature. 2015; 526 (7571): 68–74. https://doi.org/10.1038/nature15393.
44. Miller D.T., Adam M.P., Aradhya S., et al. Consensus statement: chromosomal microarray is a first-tier clinical diagnostic test for individuals with developmental disabilities or congenital anomalies. Am J Hum Genet. 2010; 86 (5): 749–64. https://doi.org/10.1016/j.ajhg.2010.04.006.
45. Bahlo M., Bennett M.F., Degorski P., et al. Recent advances in the detection of repeat expansions with shortread nextgeneration sequencing. F1000Res. 2018; 7: F1000 Faculty Rev-736. https://doi.org/10.12688/f1000research.13980.1.
46. Zimmern V., Minassian B., Korff C.A. Review of targeted therapies for monogenic epilepsy syndromes. Front Neurol. 2022; 13: 829116. https://doi.org/10.3389/fneur.2022.829116.
Об авторе
Т. В. КожановаРоссия
Кожанова Татьяна Викторовна – к.м.н., доцент кафедры неврологии, нейрохирургии и медицинской генетики педиатрического факультета; врач лабораторный генетик, ведущий научный сотрудник генетической группы научного отдела
ул. Островитянова, д. 1, Москва 117997
ул. Авиаторов, д. 38, Москва 119620
Рецензия
Для цитирования:
Кожанова Т.В. Возможности и достижения использования массового параллельного секвенирования в диагностике наследственных заболеваний с поражением нервной системы. Эпилепсия и пароксизмальные состояния. 2023;15(1):44-52. https://doi.org/10.17749/2077-8333/epi.par.con.2023.127
For citation:
Kozhanova T.V. Opportunities and achievements of using massive parallel sequencing in the diagnosis of neurodevelopmental diseases. Epilepsy and paroxysmal conditions. 2023;15(1):44-52. (In Russ.) https://doi.org/10.17749/2077-8333/epi.par.con.2023.127
JATS XML

Контент доступен под лицензией Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License.










































